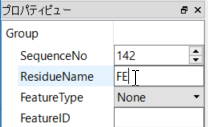
HEMEの処理について
検証環境
- 検証日:
2016/12/07
- 検証Version:
MF myPresto v3.2
概要
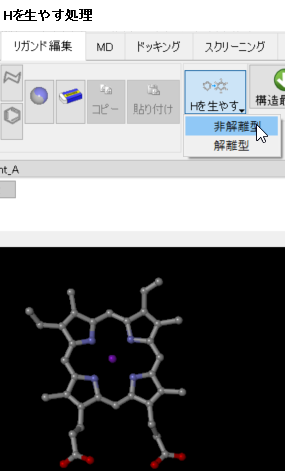
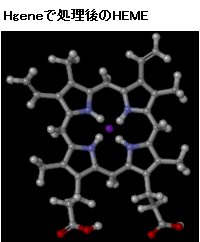
myPrestoのトポロジーファイルを作る処理において、HEMEなど、化合物が金属イオンを含む場合、化合物と金属イオンを別々のデータに分けて処理する必要があります。 myPrestoにはトポロジーファイルを作るプログラムとして、アミノ酸/核酸/金属イオン/水を処理するtplgeneと、それ以外の化合物を処理するtplgeneLというプログラムがありますが、金属イオンはtplgeneで処理して、化合物はtplgeneLで処理する必要があるためです。 また、MF myPrestoではバックグラウンドでmyPrestoのHgeneを使い、化合物に水素をつけています。化合物によっては稀に誤ったHが付く場合がありますので、その場合は水素の位置・結合を手作業で直す必要があります。 1buw.pdbでHEMEのみを抽出したデータを使い、以下処理手順を説明します。
手順
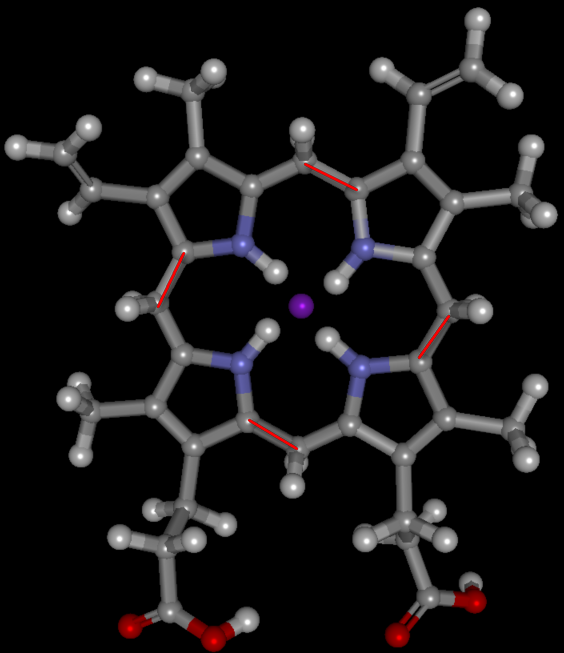
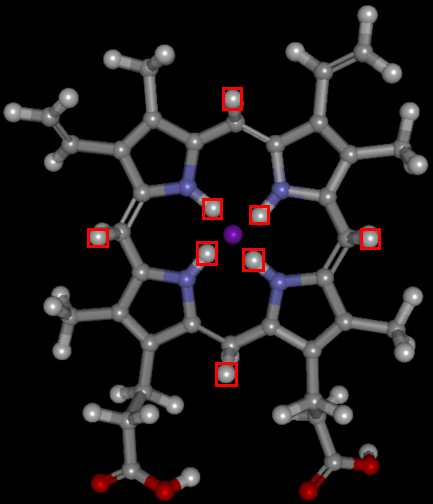
手順6で正しい構造は下記URLのとおりです。[リガンド編集]タブの[二重結合]のボタンをONにして結合を修正します。下図赤線の結合をなぞり(結合している重原子から重原子までをドラッグし)結合次数を変更します。
http://ligand-expo.rcsb.org/pyapps/ldHandler.py?formid=cc-index-search&operation=ccid&target=HEM