スクリーニング
この項の概要
この項ではスクリーニング機能の操作手順について説明します。
Note
スクリーニング機能には計算用のLinux PCが必要です。また、Torque等のジョブ管理システムと、スクリーニング用の分子データ(産業技術総合研究所が配布している下図のデータ)のインストールが必要になります。
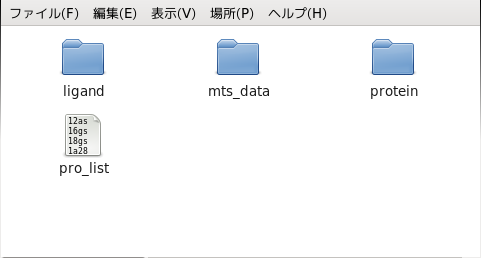
図1:スクリーニング用の分子データ
実行手順は下記の通りです。
トポロジーファイルとプローブ点を作成した分子データを用意します。
Note
DSI法で計算を行う場合、当手順は不要です。
[スクリーニング]ボタンをクリックします。
図2:[スクリーニング]のクリック
計算用PCに接続するためのSSHの設定を入力後、[既存の作業領域を開く]または[新しい作業領域を作成する]を選び、[検索]または[作成]ボタンをクリックします。
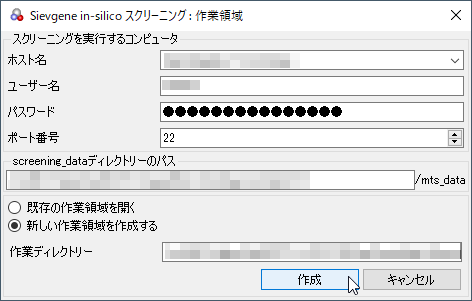
図3:[作成]のクリック
Note
計算途中でMF myPrestoを終了する場合、Workspaceファイルとして保存しておくと、次回起動時に計算途中から復帰できます。
[タンパク質のリストの準備]タブが表示されますので、[追加]ボタンからタンパク質を追加し、[次へ]ボタンをクリックします。
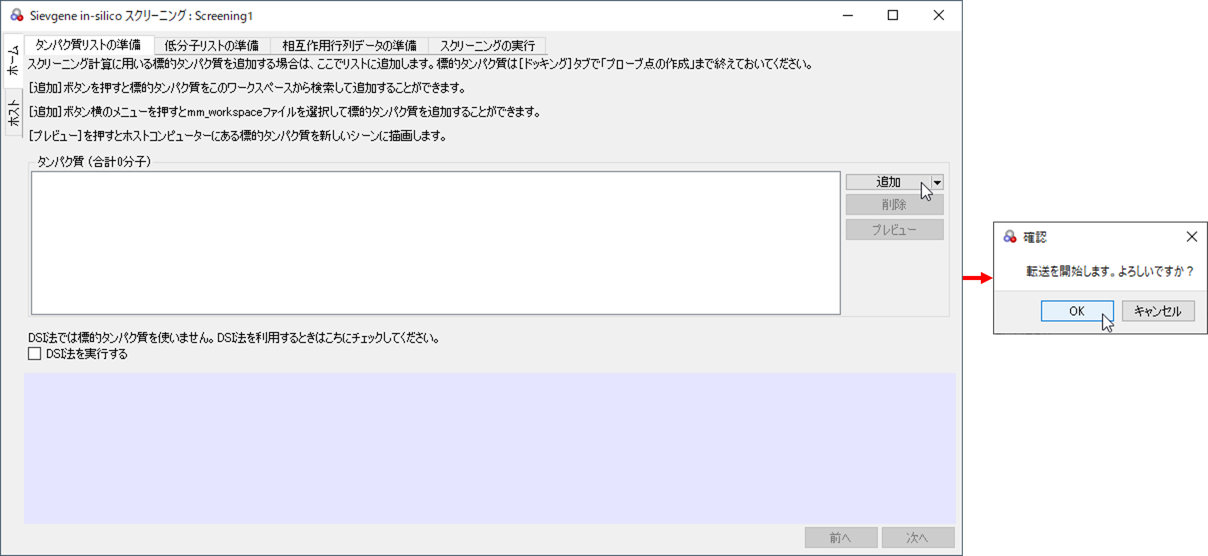
図4:[追加]のクリック
Note
ジョブ管理システムの種類を選択する場合は、上図の画面で[ホスト]タブから行います。デフォルトではLSF/OpenLavaを使うようになっています。
Note
DSI法の場合は[DSI法を実行する]にチェックします。ワークスペースに同じ名前の分子がある場合、エラーになりますので、エラーが出る場合は分子の名前をプロパティビューなどで変更してください。
[低分子リストの準備]タブが表示されますので、既存の候補化合物や機械学習用の既知活性化合物がある場合は[追加]ボタンをクリックします。[リガンドグループの追加]ダイアログが表示されますので[候補化合物][既知活性化合物]のいずれかをチェックし、[追加]ボタンからmol2ファイルのあるフォルダを選びます。設定後に[次へ]ボタンをクリックします。
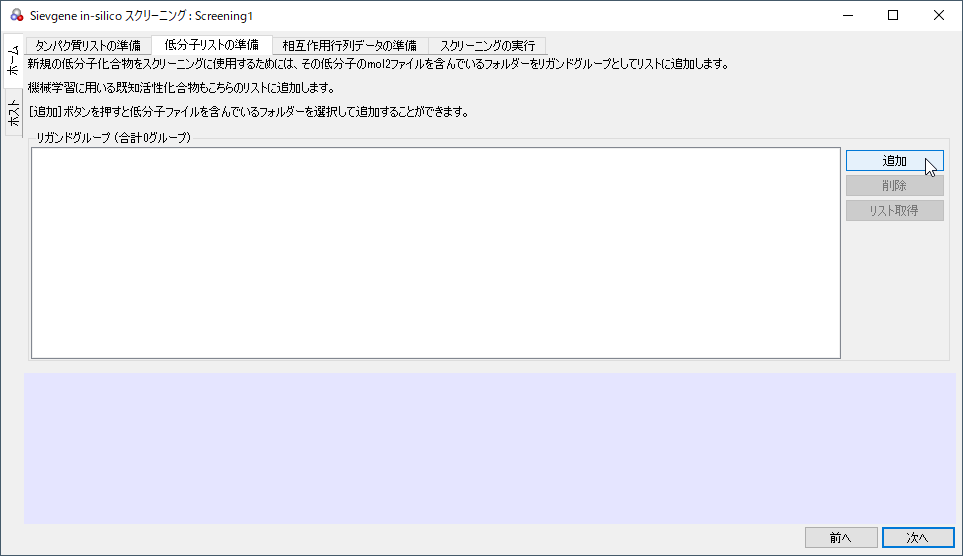
図5:[リガンドグループの追加]の[追加]ボタンでmol2ファイルがあるフォルダを指定する例
Note
[リガンドグループの追加]ダイアログの[グループ名]には任意の名前を設定します。
[相互作用行列データの準備]タブで、[新規スコアの計算]→[開始]の順にボタンをクリックすると相互作用行列の計算を開始します。
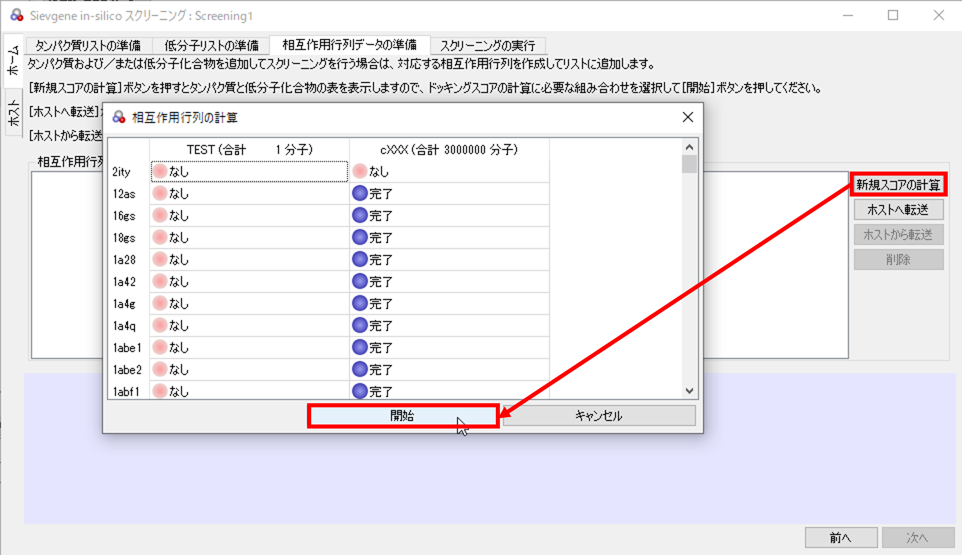
図6:[開始]のクリック
Note
計算にはしばらく時間がかかります。計算終了後は自動で[スクリーニングの実行]のタブに遷移します。
[スクリーニング実行]タブでパラメータを設定し、[スクリーニングの実行]ボタンをクリックします。
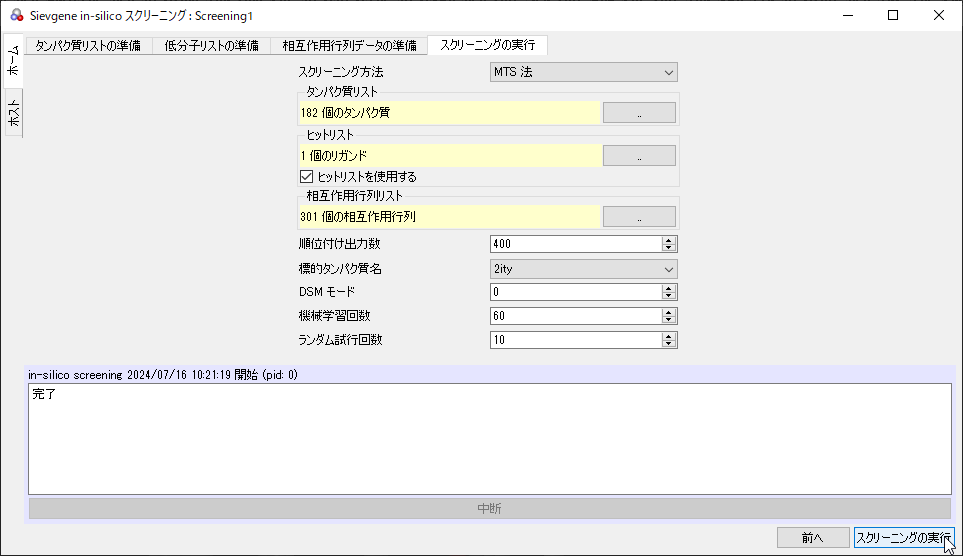
図7:[スクリーニング実行]のクリック
Note
計算にはしばらく時間がかかります。計算終了後は[スクリーニング結果]のダイアログが表示されます。
[スクリーニング結果]のダイアログが非表示の場合は[スクリーニング]タブの[結果]のボタンから表示できます。[スクリーニング結果]のダイアログのリストで項目を選択し、
 のボタンをクリックすると、構造と化合物のIDをワークスペースに読み込みます。
のボタンをクリックすると、構造と化合物のIDをワークスペースに読み込みます。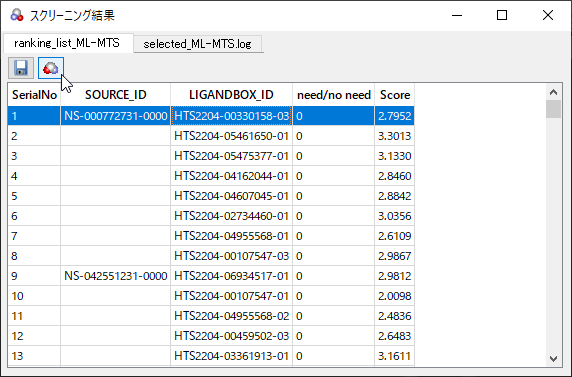
図8:スクリーニング結果のダイアログの例
Note
化合物のIDを使ってナミキ商事やキシダ化学から実際の試薬を購入できます。